From 生物技能树 GEO数据挖掘第二节(Day9接Day10前半节)
探针注释
注意:只有基因表达芯片数据才需要找探针注释
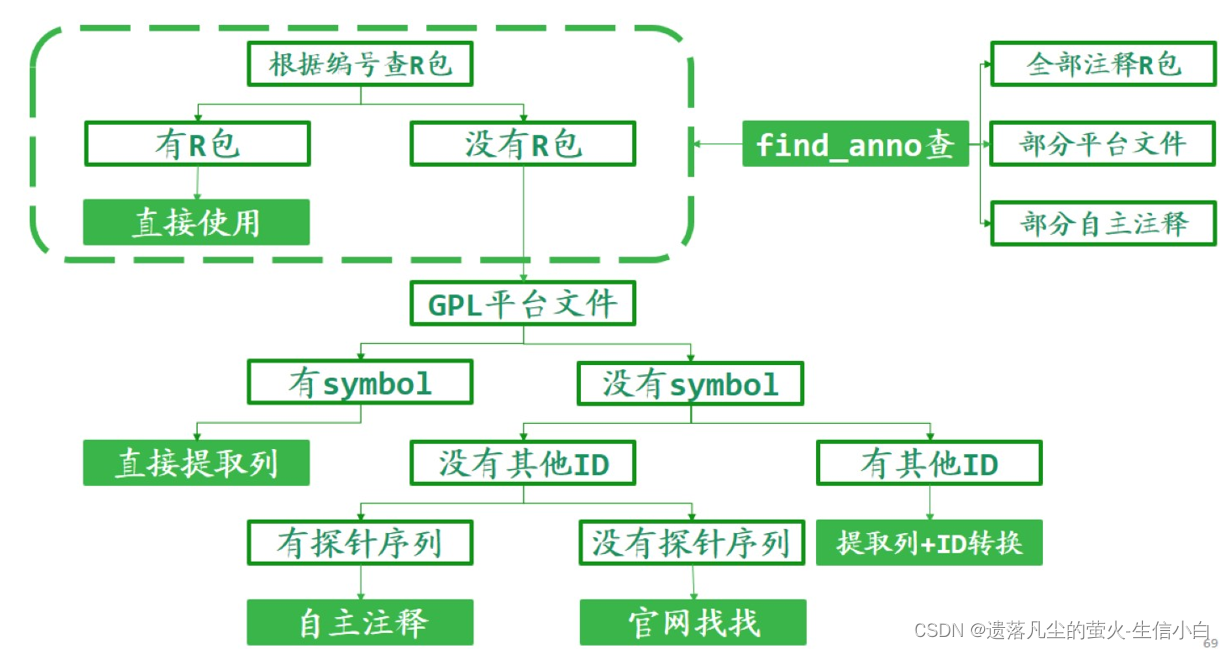
自主注释流程(了解)
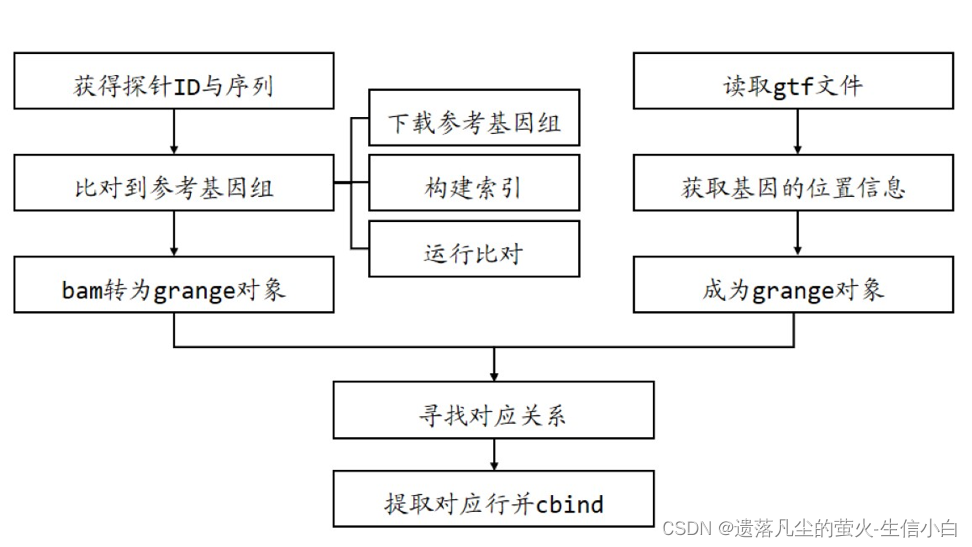
探针注释示例数据
:GSE2378
该数据具有两个表达矩阵文件,看GPL

rm(list = ls()) #打破下载时间的限制,改前60秒,改后10w秒 options(timeout = 100000) options(scipen = 20)#不要以科学计数法表示 #传统下载方式 library(GEOquery) eSet = getGEO("GSE2378", destdir = '.', getGPL = F) #网速太慢,下不下来怎么办 #1.从网页上下载/发链接让别人帮忙下,放在工作目录里 #2.试试geoChina,只能下载2019年前的表达芯片数据 #library(AnnoProbe) #eSet = geoChina("GSE2378") #选择性代替第8行 #研究一下这个eSet class(eSet) length(eSet) eSet = eSet[[1]] class(eSet) #(1)提取表达矩阵exp exp <- exprs(eSet) dim(exp) range(exp)#看数据范围决定是否需要log,是否有负值,异常值 exp = log2(exp+1) #需要log才log boxplot(exp,las = 2) #看是否有异常样本 exp = limma::normalizeBetweenArrays(exp) #如果有异常值可以调平 #(2)提取临床信息 pd <- pData(eSet) #查看pd,查看分组信息,分组在description这一列,用pd$description查看具体信息 #(3)让exp列名与pd的行名顺序完全一致 p = identical(rownames(pd),colnames(exp));p if(!p) { s = intersect(rownames(pd),colnames(exp)) exp = exp[,s] pd = pd[s,] } #(4)提取芯片平台编号,后面要根据它来找探针注释 gpl_number <- eSet@annotation;gpl_number save(pd,exp,gpl_number,file = "step1output.Rdata") # 原始数据处理的代码,按需学习 # https: # Group(实验分组)和ids(探针注释) rm(list = ls()) load(file = "step1output.Rdata") # 1.Group---- library(stringr) # 标准流程代码是二分组,多分组数据的分析后面另讲 # 生成Group向量的三种常规方法,三选一,选谁就把第几个逻辑值写成T,另外两个为F。如果三种办法都不适用,可以继续往后写else if if(F){ # 第一种方法,有现成的可以用来分组的列 }else if(F){ # 第二种方法,眼睛数,自己生成 Group = rep(c("Disease","Normal"),each = 10) # rep函数的其他用法?相间、两组的数量不同? }else if(T){ # 第三种方法,使用字符串处理的函数获取分组 k = str_detect(pd$title,"N");table(k) Group = ifelse(k,"Normal","Disease") } # 需要把Group转换成因子,并设置参考水平,指定levels,对照组在前,处理组在后 Group = factor(Group,levels = c("Normal","Disease")) Group data.frame(pd$title,Group) #检查一下分组是否正确 #2.探针注释的获取----------------- #捷径 library(tinyarray) find_anno(gpl_number) #辅助写出找注释的代码 library(hgu95av2.db);ids <- toTable(hgu95av2SYMBOL) #BiocManager::install("hgu95av2.db") 安装R包 #这是从28行运行结果里复制下来的代码👆 #如果能打出代码就不需要再管其他方法。 #如果使用复制下来的AnnoProbe::idmap('xxx')代码发现报错了,请注意尝试不同的type参数 #如果显示no annotation avliable in Bioconductor and AnnoProbe则要去GEO网页上看GPL表格里找啦。 #四种方法,方法1里找不到就从方法2找,以此类推。 #捷径里面包含了全部的R包、一部分表格、一部分自主注释 #方法1 BioconductorR包(最常用,已全部收入find_anno里面,不用看啦) if(F){ gpl_number #看看编号是多少 #http: library(hgu133plus2.db) ls("package:hgu133plus2.db") #列出R包里都有啥 ids <- toTable(hgu133plus2SYMBOL) #把R包里的注释表格变成数据框 } # 方法2 读取GPL网页的表格文件,按列取子集 ##https: # 方法3 官网下载注释文件并读取 # 方法4 自主注释,了解一下 #https: save(exp,Group,ids,file = "step2output.Rdata") 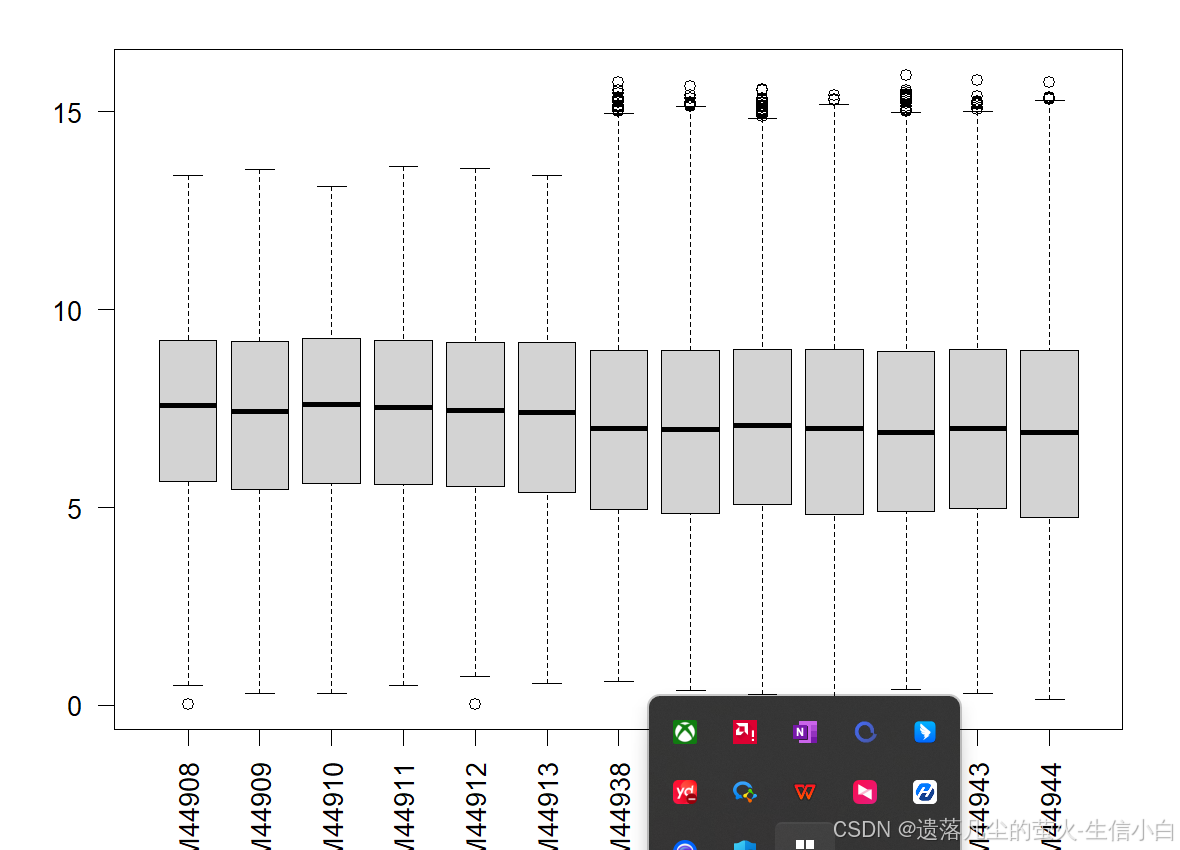
# 鉴于出来的PCA显示数据具有批次效应,只选取前六个样品进行分析 rm(list = ls()) #打破下载时间的限制,改前60秒,改后10w秒 options(timeout = 100000) options(scipen = 20)#不要以科学计数法表示 #传统下载方式 library(GEOquery) eSet = getGEO("GSE2378", destdir = '.', getGPL = F) #网速太慢,下不下来怎么办 #1.从网页上下载/发链接让别人帮忙下,放在工作目录里 #2.试试geoChina,只能下载2019年前的表达芯片数据 原文链接:https://blog.csdn.net/weixin_57975238/article/details/139012671?ops_request_misc=%257B%2522request%255Fid%2522%253A%2522786c59bd3d88903e1920cd5e80270d4b%2522%252C%2522scm%2522%253A%252220140713.130102334.pc%255Fblog.%2522%257D&request_id=786c59bd3d88903e1920cd5e80270d4b&biz_id=0&utm_medium=distribute.pc_search_result.none-task-blog-2~blog~first_rank_ecpm_v1~times_rank-12-139012671-null-null.nonecase&utm_term=GEO
© 版权声明
文章版权归作者所有,未经允许请勿转载。
相关文章

暂无评论...