1.生存分析与差异分析
首先在进行数据处理之前,我们需要知道做生存分析和差异分析都需要些什么类型的原始数据。
①差异分析:精准找出在两组 / 多组样本间表达水平存在统计学显著差异的分子,如差异表达基因(DEGs)。需要的输入数据是不同基因在不同样本里面的表达矩阵,需要包含的是正常样本和肿瘤样本。
②生存分析:通过生物信息学工具和统计模型,结合基因表达、突变与 临床生存数据(如生存时间、是否发生终点事件),挖掘影响样本(通常是患者)生存预后的基因,并评估其预后预测价值的核心分析手段。需要输入的数据不同肿瘤样本的表达基因。
所以如果需要做生存分析和差异基因就需要从GEO数据库获取表达矩阵和生存信息,但是并不是所有的数据集都有生存数据,所以没有生存数据的数据集只可以用来进行差异分析;只有肿瘤样本的数据集不可以使用来做差异分析。这些都可以在GEO的数据集详情页看到。
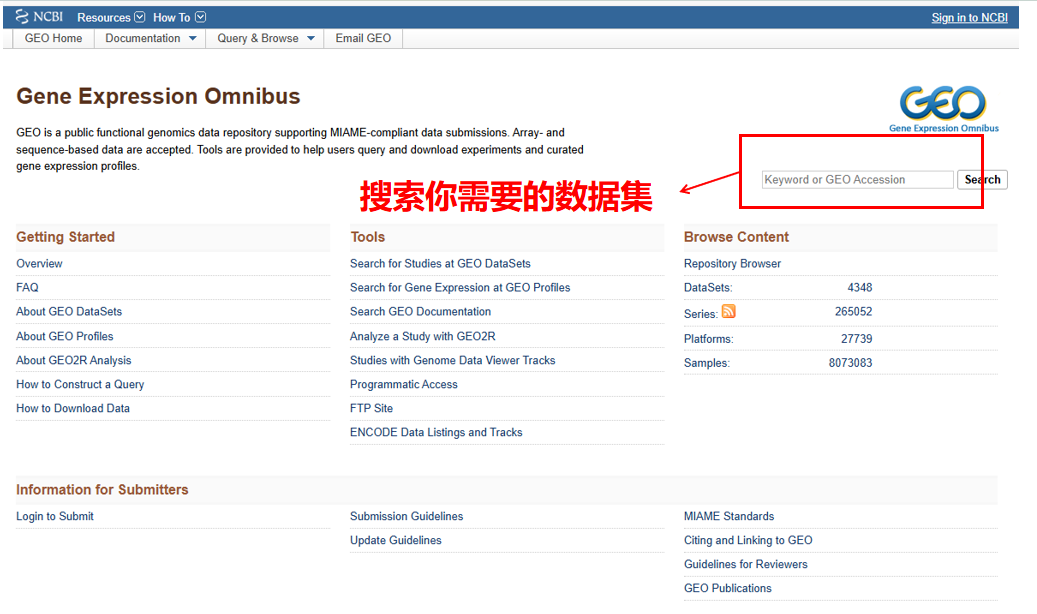
2.GEO数据获取与处理
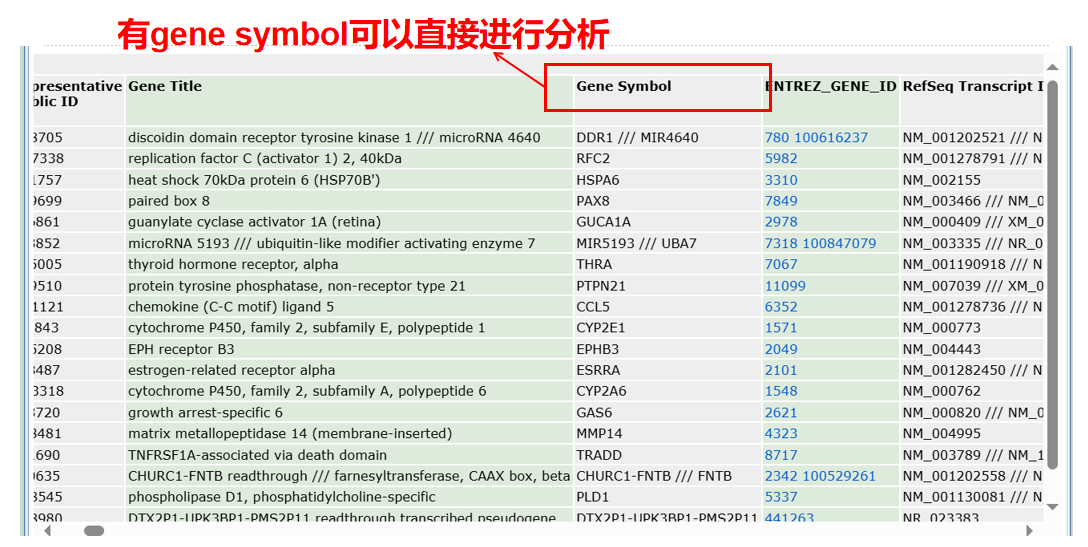
①处理得到EXP文件:我们首先需要从GEO数据库获取表达矩阵文件,另一个是平台注释文件。因为GEO数据库里面的表达矩阵使用的是基因探针ID,我们是不知道它到底是哪一个基因,所以就需要平台注释文件对每一个探针ID转换成gene symbol。使用R脚本将ID转换成symbol之后得到EXP文件就可以进行后续的差异基因分析。但是并不是所有的平台文件都有对应gene symbol,因为有一些数据集会被加密,这些数据库就是没有对应相应的gene symbol,这种数据库要么需要破译要么只能放弃。
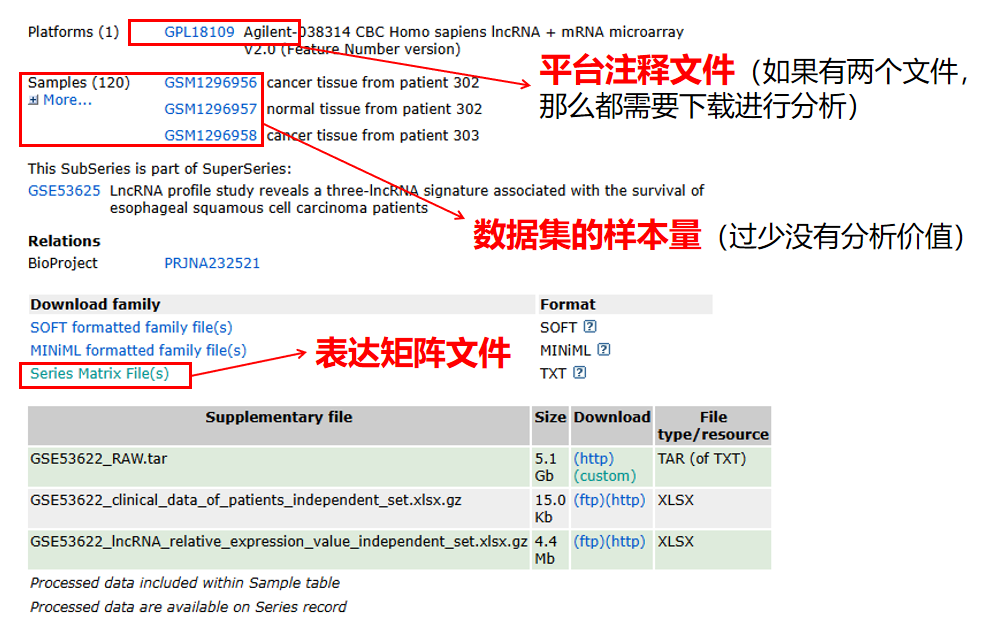
在点开平台注释文件之后我们可以看到每一个ID是否有对应gene symbol:

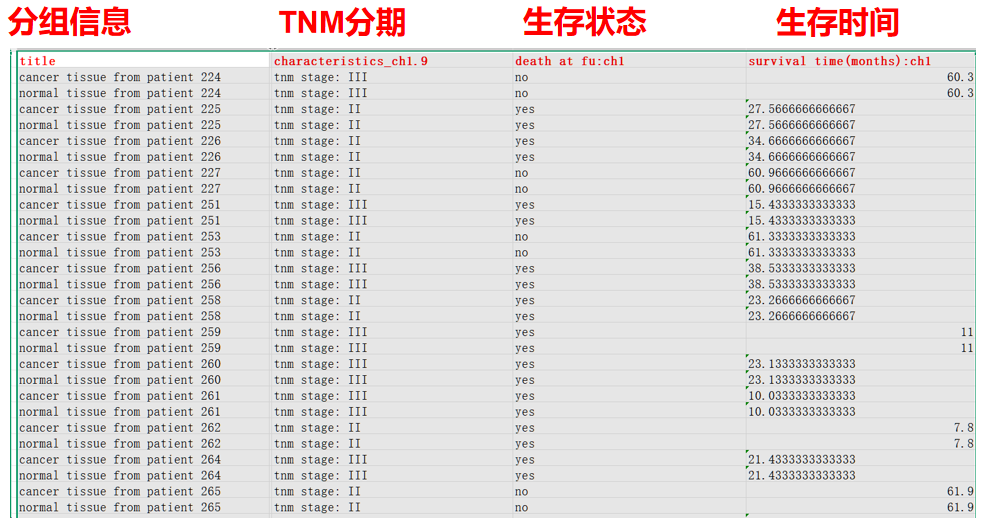
②处理得到Clinical文件(方便进行分组进行–差异分析;同时提取临床分期情况–生存分析):在表达矩阵的前面会有样本的临床信息:生存时间、分期等等,这时就需要使用R语言里面的pd=pData()命令提取 ExpressionSet 对象的“样本-水平”信息表(phenotype data,即临床/实验条件、性别、年龄、分组等),最后就可以得到用于后续分析的Clinical文件。
在得到的ExpressionSet 对象的“样本-水平”信息表里面需要找到下面这几列
#不是所有的信息表都有这些数据,有些数据表里面只有分组信息,只有分组信息的就只能做差异;做差异如果不需要细致到对每一个分期都进行分析的话就不需要查找TNM分期数据;做生存分析就一定需要生存状态与生存时间,注意生存时间的转换,这里的生存时间单位是月,一般做生存是要用年,所以后续需要注意转换。
同时注意跑生存的时候,只需要肿瘤样本,注意将正常样本去除!
所以最后可以通过跑脚本得到两个文件:EXP(表达矩阵文件)、Clinical文件(临床文件)去2进行后续的差异以及生存分析。
最后,注意GEO有些数据格式是count,有些是TPM格式,所以需要进行一个简单的判断之后看看需不需要进行转换,可以通过以下代码进行简单的判断与转换。

相关文章




