博主最近在做机器学习相关,需要用到大量的数据,这就需要在GEO上频繁下载数据库(被折磨得神志不清),所以就简单做了一个快速整理geo数据库的R脚本,分享给大家。
核心思路很简单,就是利用GEOquery包中的在线下载注释的功能,随后将所有的变量名称通过文件夹名称和txt名称来进行替换,进而解放双手。
1、前置准备
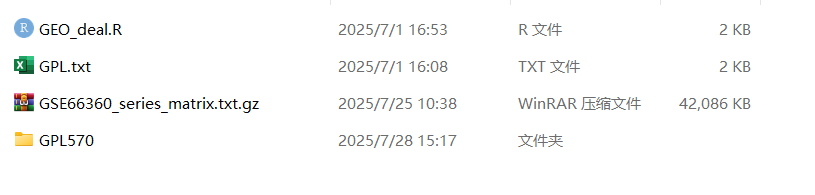
新建一个文件夹放入准备内容,文件夹的名称为该芯片的名称,准备的内容为:R脚本、GPL.txt,里面包含的是所有拥有在线注释的GPL平台名称、在GEO中下载的压缩包、以及新建一个空白文件夹,文件夹的名称为该芯片的GPL平台。
2、代码部分
2.1、包的载入
# GEO数据整理 library(GEOquery) library(dplyr) library(ggplot2) ##3.5.0 library(ggpubr) ##0.6.0 library(reshape2) ##1.4.4 library(here) library(fs) current_path <- getwd() setwd(current_path) current_folder <- basename(getwd()) dataname <- current_folder subdirs <- dir_ls(type = "directory") platfrom <- path_file(subdirs)
这里的getwd()函数的功能是获取当前R脚本所在的路径,所以使用前只需要我们把R脚本放到相应的文件夹就可以;dir_ls()是获取当前目录下所有的子文件夹的名称,然后通过path_file()提取最后一个文件夹的名称
2.2、代码主体
if(platfrom %in% rownames(GPL) == TRUE){ lib1 <- GPL[rownames(GPL) == platfrom,] lib <- paste0(lib1,".db") library(lib,character.only = T) gset <- getGEO(dataname, destdir = current_path,AnnotGPL = F,getGPL = F) dat <-exprs(gset[[1]]) exprt <- t(dat) #获取临床数据/导出临床数据 phe <- gset[[paste0(dataname,"_series_matrix.txt.gz")]]@phenoData@data write.table(data.frame(ID=rownames(phe),phe),file = "C_GSE.txt",sep = "\t",quote = F,row.names = F) ls(paste0("package:",lib)) ids <- toTable(get(paste0(lib1,"SYMBOL"))) colnames(ids) <- c("probe_id", "symbol") # 平台文件的ID和矩阵中的ID匹配。%in%用于判断是否匹配。 ids=ids[ids$probe_id %in% rownames(dat),] # 获取匹配的表达数据 dat=dat[ids$probe_id,] # 查看探针名与注释文件名是否完全一致 table(rownames(dat) == ids$probe_id) # 合并 dat <- cbind(ids, dat) # 删除重复基因,保留最大值 dat <- aggregate(. ~ symbol, data=dat, max) rownames(dat) <- dat$symbol expr <- dat[,-c(2,2)] write.table(expr,file = "Exp.txt",sep = "\t",row.names = F,quote = F) }else{ wrong <- "未找到匹配的GPL注释文件" writeLines(wrong, "wrong.txt") }
需要注意的是,如果在运行过程中出现报错:不存在叫‘xxx.db’这个名称的程序包

则说明没有安装相应的注释包,可以使用以下代码进行安装,安装后重新运行代码即可:
BiocManager::install(lib,character.only = T)
当然,如果你先麻烦,可以直接写一个循环把所有GPL中所有的包都安装上。这里就不再展示(不过鉴于R语言的下载速度,有很大概率会失败😢)
3、打包封装
同时博主也用python封装了一个exe,原理就是上述的R语言代码,只不过为了方便组内同学(和我)使用,制作了用户界面,同时加装了一些小功能,使用起来会更加丝滑。
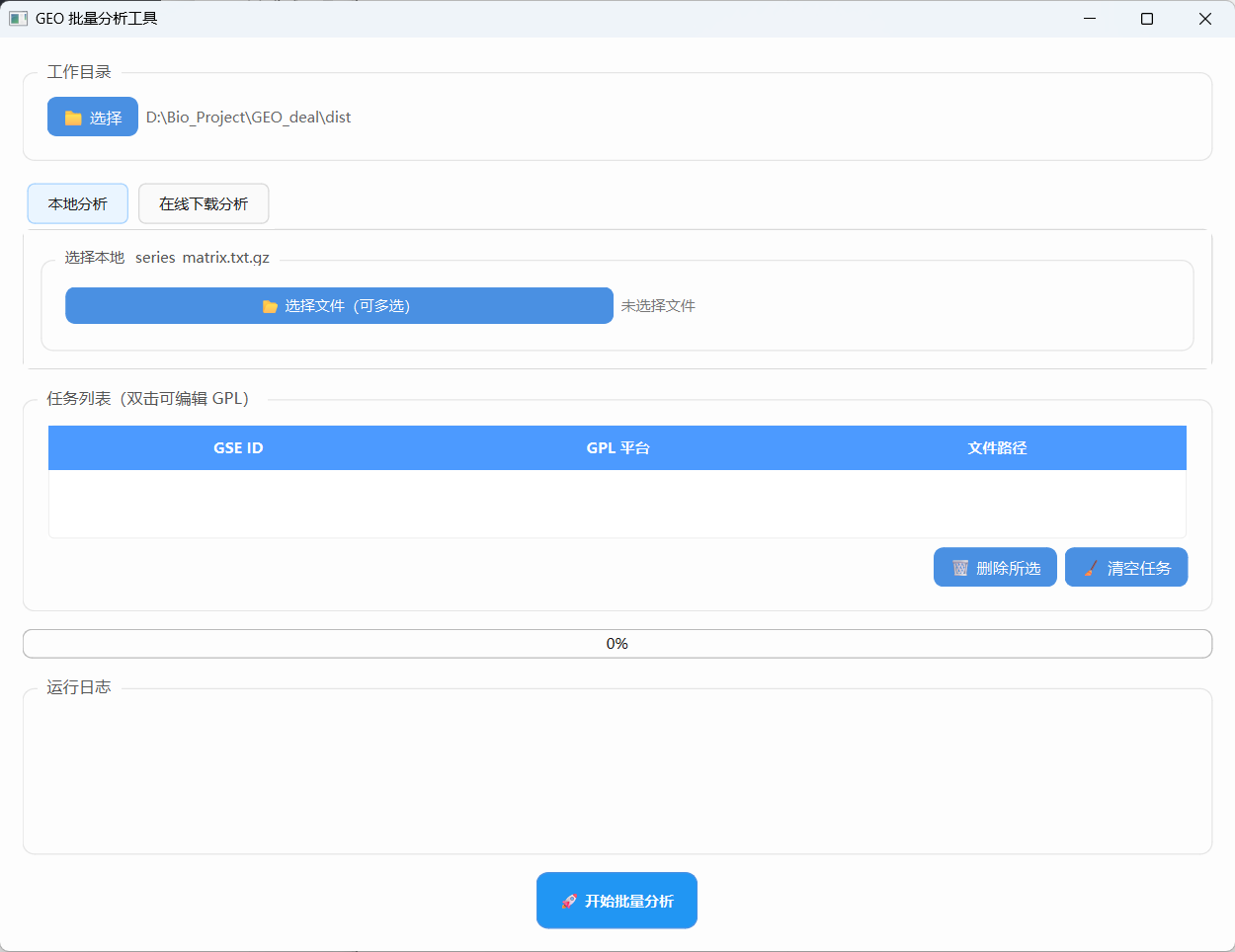
软件的使用如下:
1、选择工作目录,目录下的子文件夹为数据库名称,里面有拥有XXX_series_matrix.txt.gz数据集,点击选择文件,选中该数据集后会弹出窗口,输入GPL平台(输错了可以在任务列表中双击对应的平台进行修改)
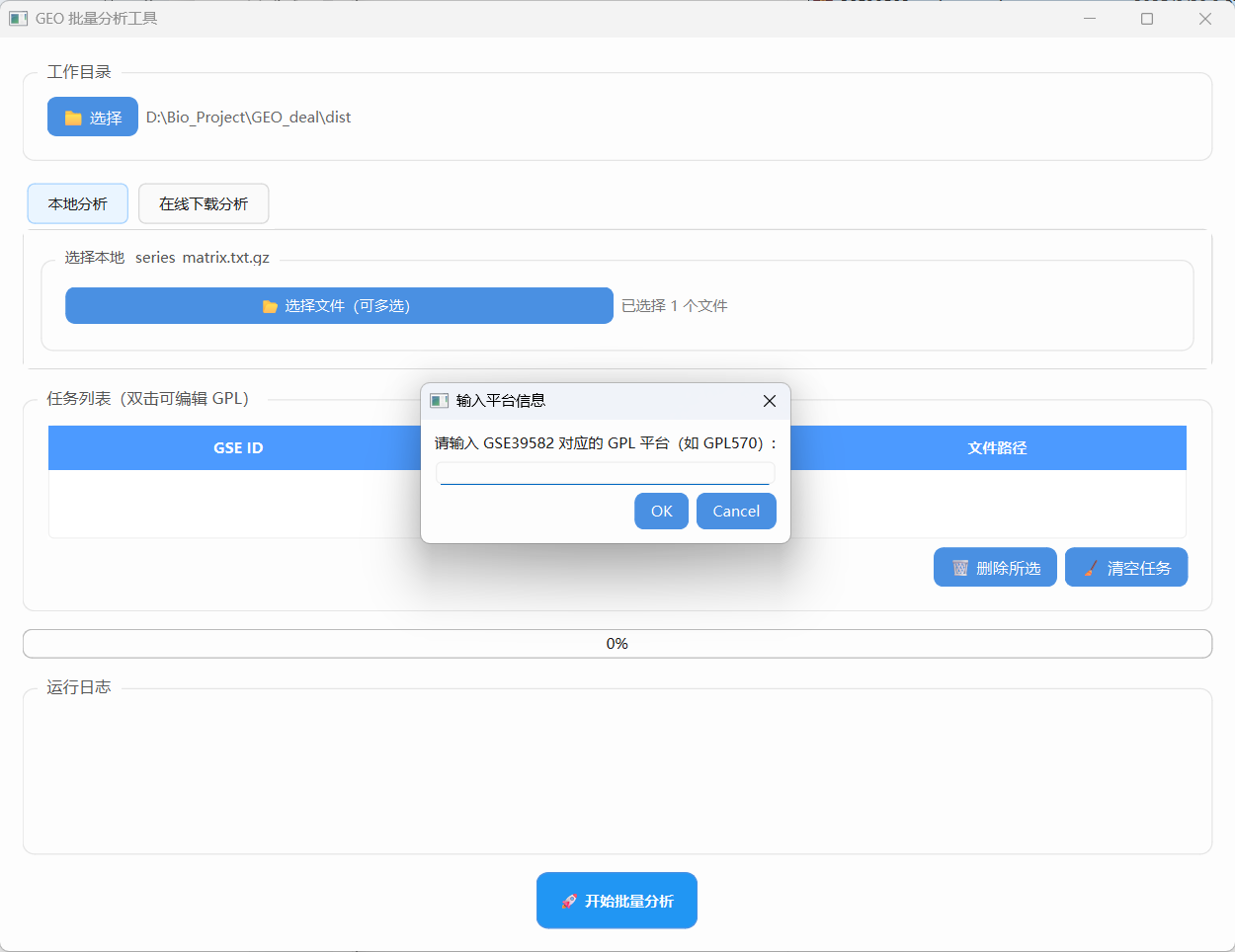
2、点击开始批量分析按钮,即可开始分析,前提时以及装好了对应平台的.db文件,否则无法输出,程序只会报对(忘记做报错了),只能在运行日志中检查错误信息
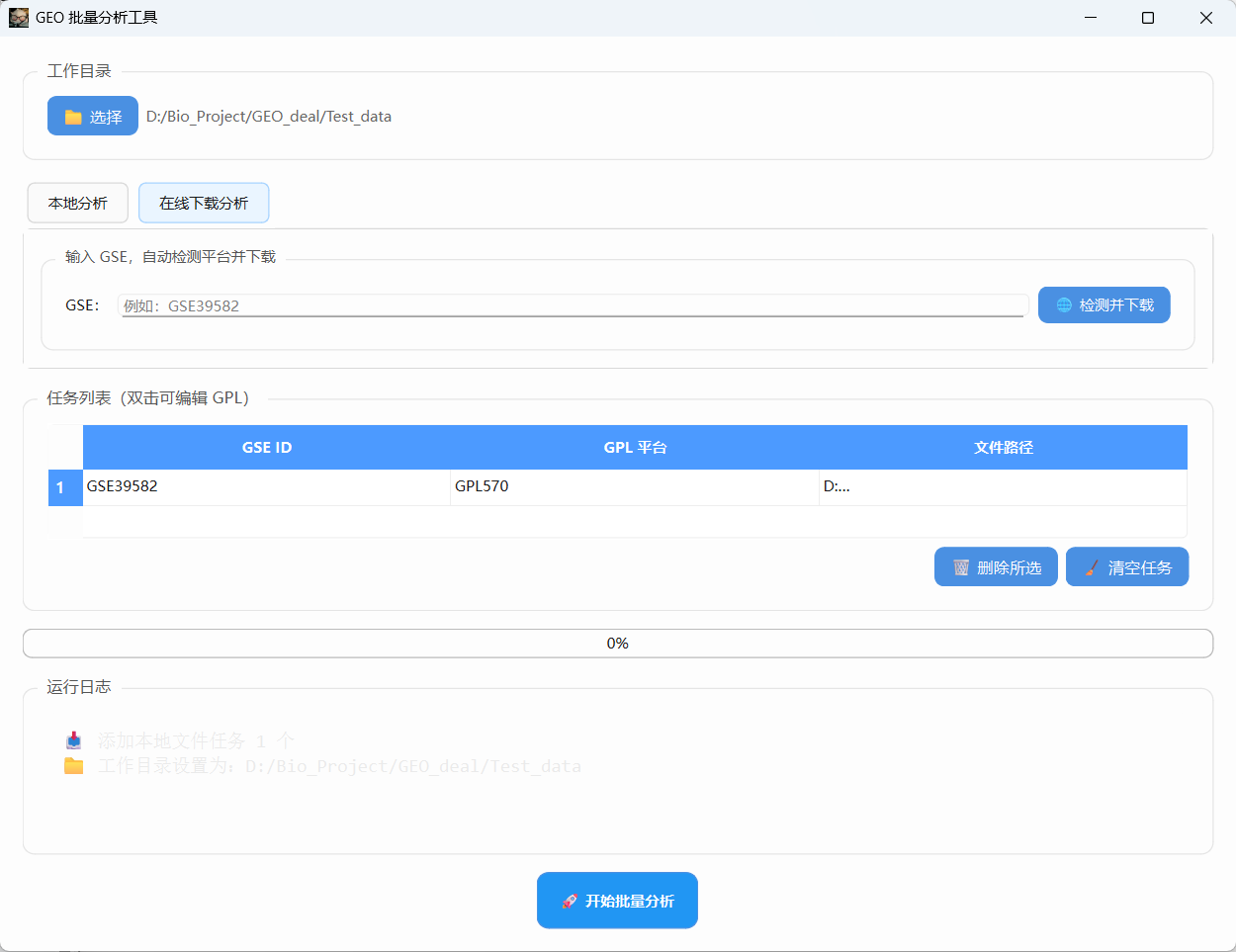
3、在线下载分析只需要输入数据集名称,就可以通过爬虫自动下载数据集(其实GEOquery包也可下载,但是R语言下载真的太慢了),并且检测平台信息(稳定性虽然比GEOquery好,但有时候可能还时不如直接用迅雷下数据集,本地分析来得快。后面有了千兆网我会用吧…战未来)。这个功能只需要选择工作目录,就会在工作目录下自动创建文件夹,下载完成后点击分析即可
软件地址:https://pan.baidu.com/s/1QNoD8ZbLZ5qIhFndp9rrmA?pwd=vrgu
提取码: vrgu
相关文章



