GEO上所有数据集数据的下载和提取的最详细教程
0. GEO数据库讲解和准备
NCBI GEO网站介绍
NCBI GEO数据库(Gene Expression Omnibus)是一个全球最大的生物医学领域的公共数据库平台,用于存储基因组学数据,包括基因表达数据、染色质状态和基因组变异等。研究人员可以在该平台上查找、下载和分析各种基因表达数据集,以便进行生物信息学和生物医学研究。NCBI GEO数据库提供了丰富的数据资源和分析工具,是生命科学领域的重要研究工具之一。大量生物医学领域发表的论文使用的公共数据集也一般是来自GEO数据库的数据集。
GEO数据库中的数据集按照一系列预定义的格式和标准进行提交和组织,主要包括以下几种格式:
1.GSE(Gene Series):表示一个或多个实验的数据集,通常包含多个样本。
2.GSM(Sample Series):表示单个实验中的一个样本。
3.GPL(Platform):描述用于实验的数据采集平台的详细信息,包括探针序列、实验条件等。
4.GDS(Series Matrix File):包含一个GSE或GSM数据集的详细矩阵表格,其中行表示基因,列表示样本,单元格中的值表示基因在样本中的表达水平。
配置aria2c高效下载支持断点续传的下载神器来加速GEO的数据下载
为了在国内也能稳定下载GEO的数据,必须要配置一下aria2c这个下载神器工具。OmicsTools的GEO数据下载模块会调用aria2c来实现在国内对美国的NCBI GEO数据库上的数据集进行文档下载和断点续传。

aria2c配置的教学视频链接

Aria2c软件下载
在D:/omics_tools目录下创建一个aria2的目录,从我的生信群里下载aria2c.exe软件包到D:/omics_tools/aria2目录下就可,不需要安装,只需要把这个aria2c.exe放到该目录就行了。
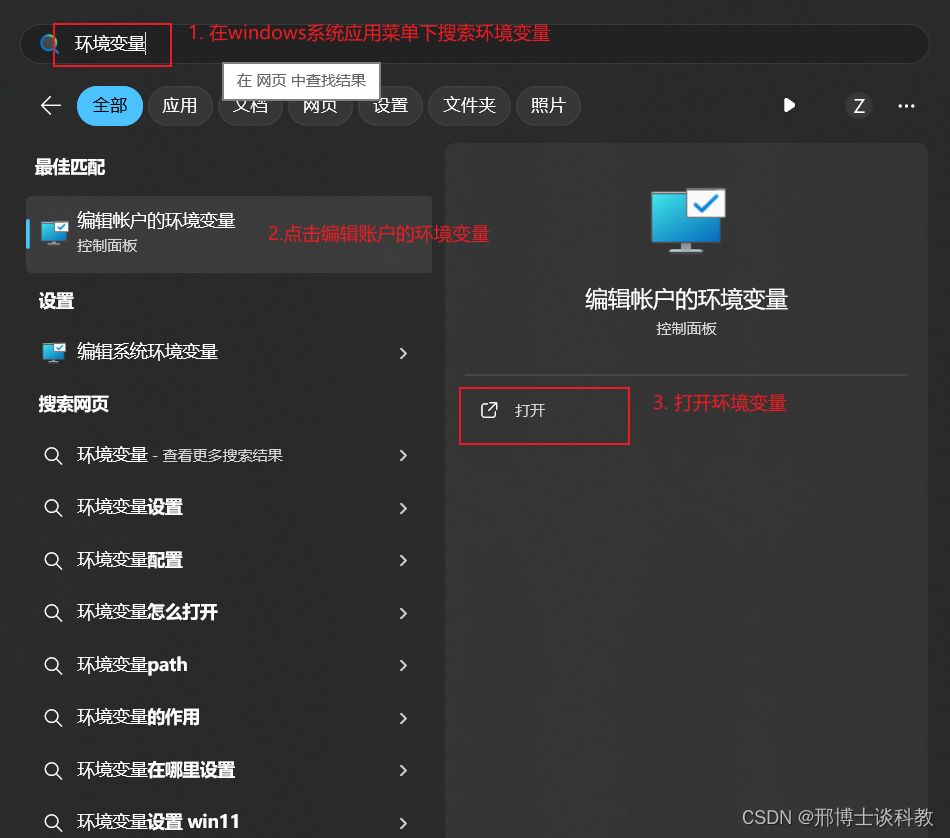
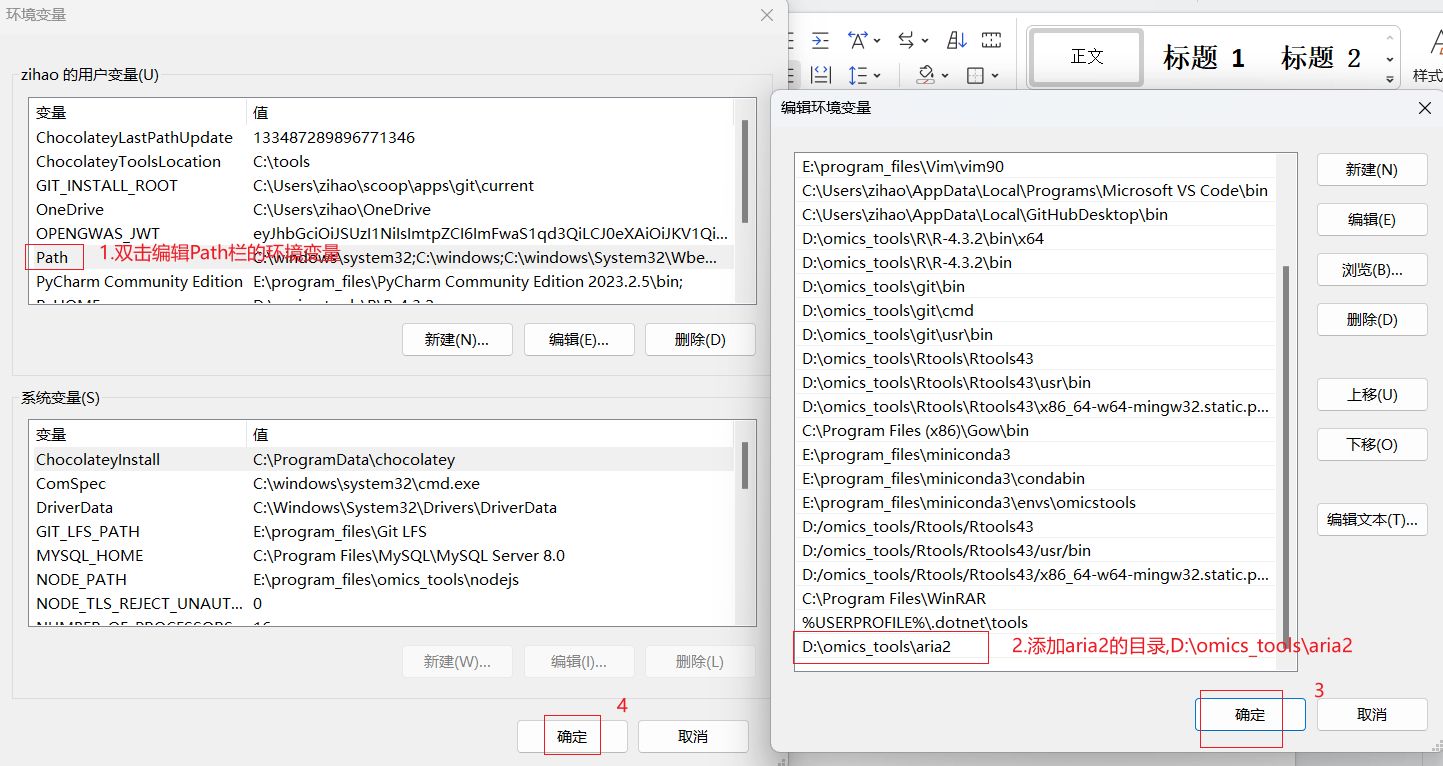
配置aria2c的环境变量
环境变量的配置示意图
检测aria2c的环境变量是否配置成功
新打开一个cmd命令提示符,输入which aria2c, 看看是否会返回aria2c的路径
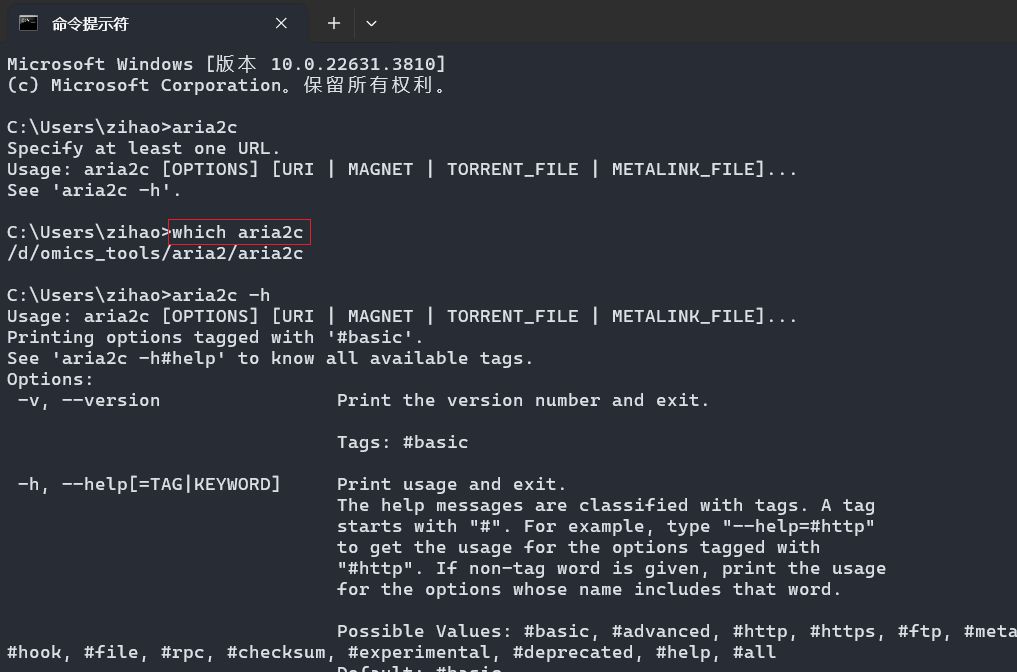
如果成功返回了aria2c的路径,至此,aria2c下载工具就配置完成了。
GEO数据下载教程
1.1根据GEO的GSE数据集编号自动下载和处理GEO数据教程(必须要运行的模块,GEO数据下载要首先运行这个模块)
该模块所在的位置和软件界面
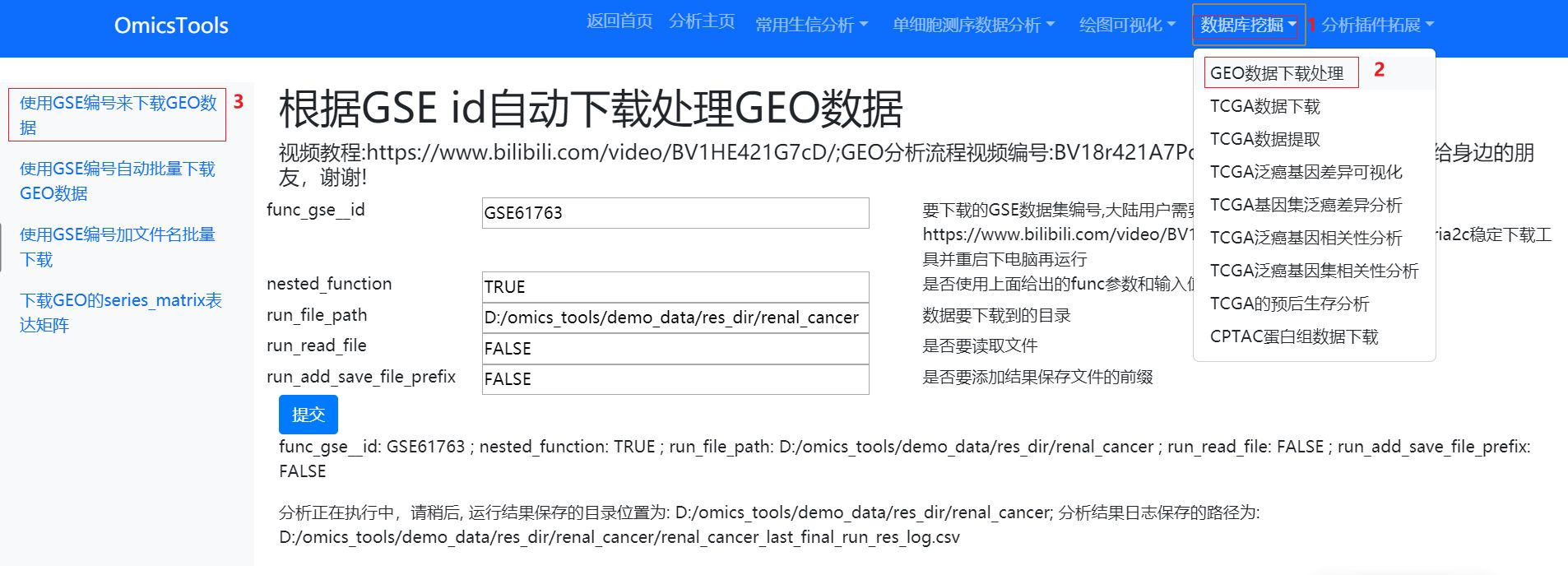
该模块的教学视频链接

运行中的显示信息
分析正在执行中,请稍后, 运行结果保存的目录位置为: D:/omics_tools/demo_data/res_dir\res_dir; 运行结果日志保存的路径为: D:/omics_tools/demo_data/res_dir\res_dir\renal_cancer_last_final_run_res_log.csv
运行完成的显示信息
执行已完成,运行结果保存的目录位置为: D:/omics_tools/demo_data/res_dir/renal_cancer; 分析结果日志保存的路径为: D:/omics_tools/demo_data/res_dir/renal_cancer\renal_cancer_last_final_run_res_log.csv
下载整理到的结果文件展示
结果文件列表
结果文件列表
样本注释文件信息
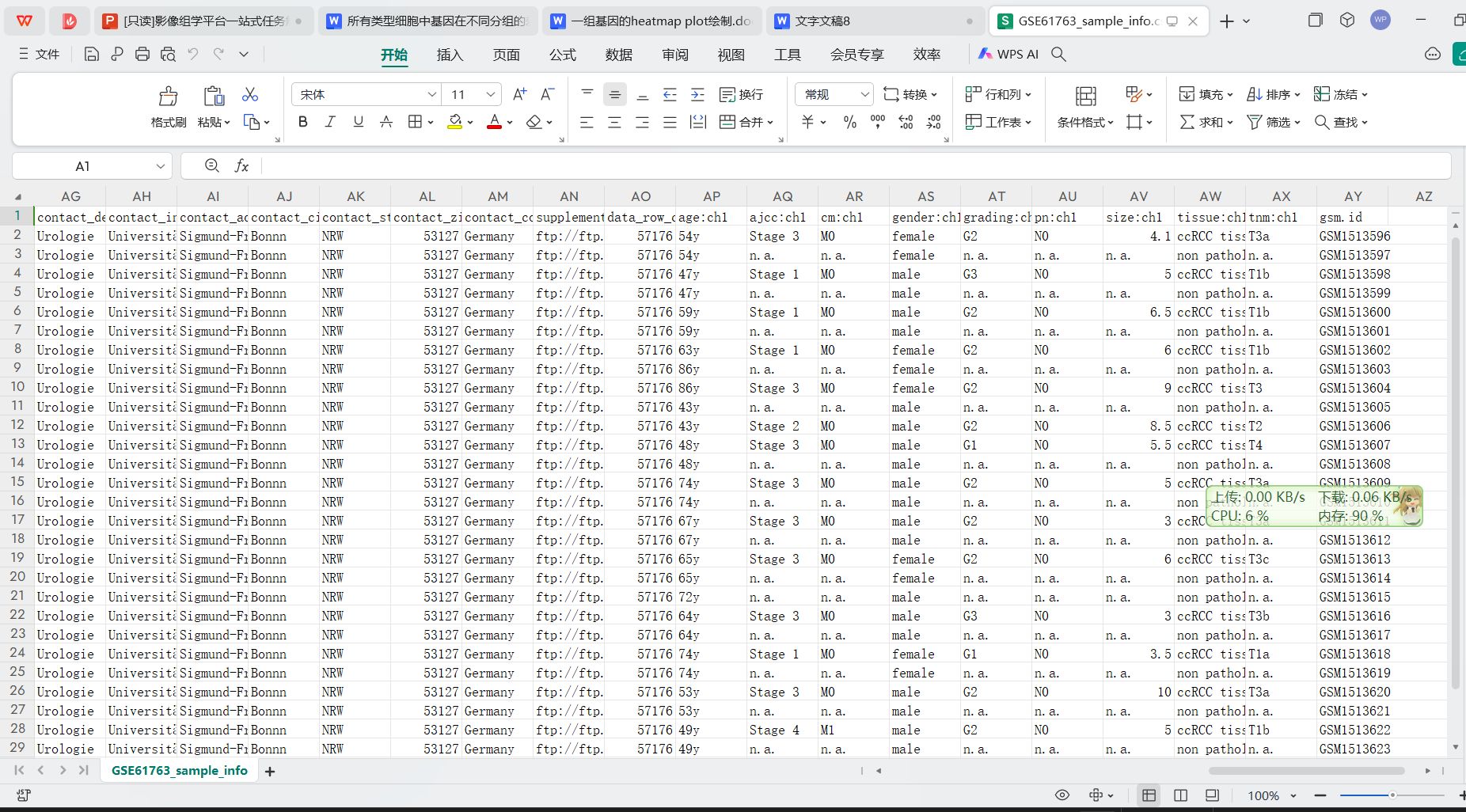
样本的注释信息文件
基因表达矩阵
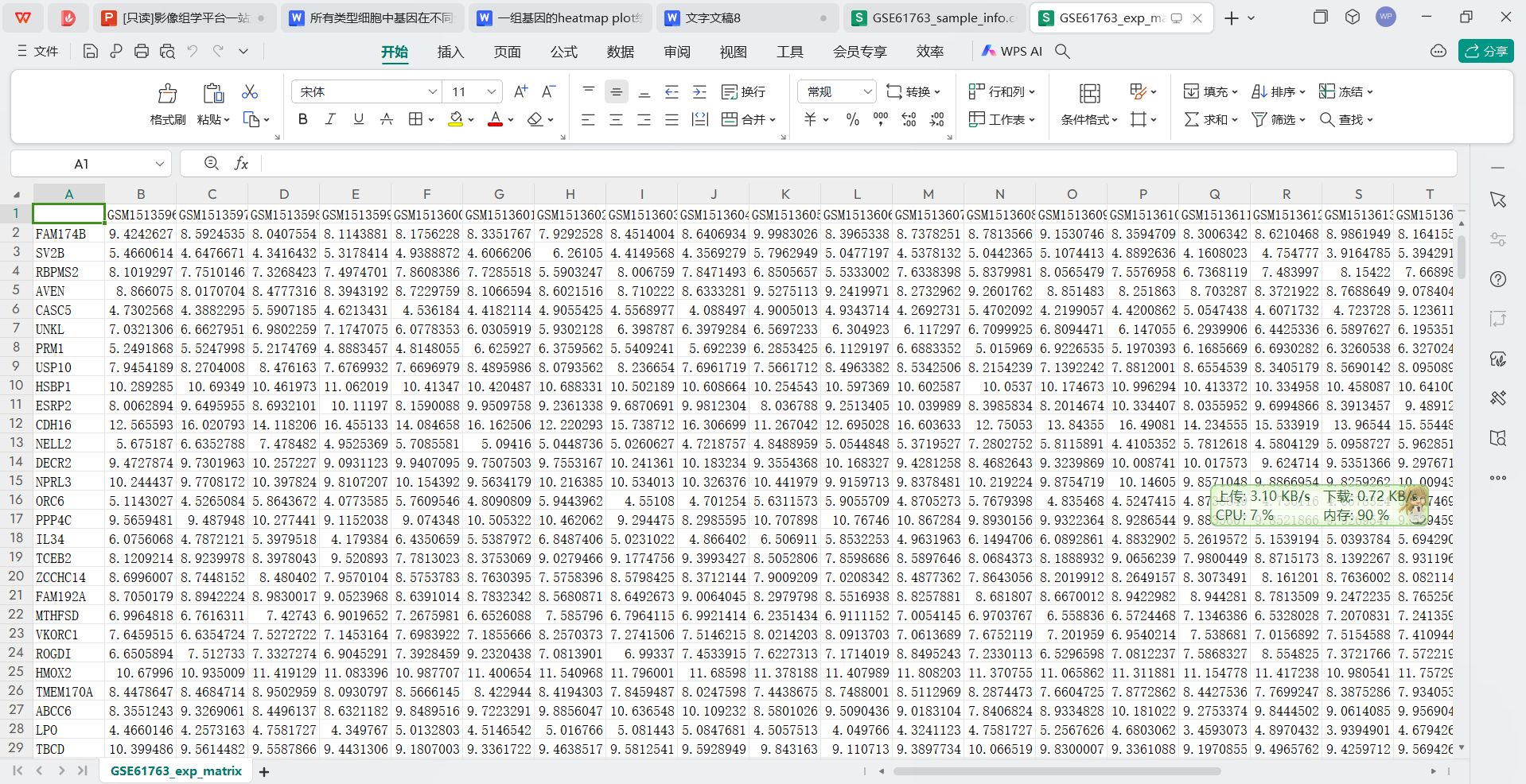
基因表达矩阵文件
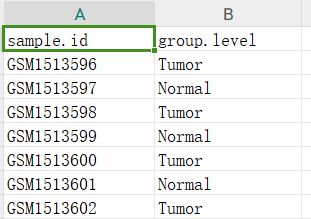
下载GEO数据一定要先运行下这个模块,部分数据集不一定能下载提取出表达矩阵,但是一般能下载到非常完整的样本的注释信息等数据文件,这在对GEO数据集临床信息分析或分组差异分析是非常重要的,当然如果少数GSE数据集遇到没有提取出GSEXXX_sample_info.csv的情况,也可以看看我b站的教学视频,根据GEO网页中的样本编号的分组情况,自建一个这样的分组文件,分组文件一般是长这个样子的,有两列,一列列名叫sample.id,是GSM编号信息,另一列是group.level,存放的是分组信息,当然sample.id这一列的样本名还是要以表达矩阵的样本名为准,这样才能让表达矩阵后面跟样本分组信息按照相同的列名整合在一起:
对于部分没有下载出表达矩阵的数据集,如果该数据集的GEO网页中有GSEXXX_RAW.tar格式的压缩包,可以运行下面的1.2这一步对RAW.tar的压缩包文件进行提取和处理。
该GEO下载模块下载不同GSE数据集得到的不同下载结果详细答疑:
GEO的数据集格式多种多样,使用该模块下载GE
相关文章




